解析 Ensembl 的数据库服务器
Ensembl 对于学习生物信息学的我们来说应该是如数家珍了,但是我们平时常用到的操作一般是去查基因,下载基因组,这些都是通过网页端来完成:http://uswest.ensembl.org/index.html
上面网页展示的就像大家进入某宝商店中的商品展示图,而真正的货物是存放在仓库的。Ensemble 则是将其“货物”是存放在数据库服务器中。
理解 Ensembl 的数据库存储结构与架构,对于我们的今后的生信研究工作大有裨益。
接下来,我们进入 Ensembl 的数据仓库世界,去看看它的货物有哪些,我们怎么来使用这些数据。
一、Ensembl 数据库服务器介绍
Ensembl 一共提供四个数据库服务器访问地址:
ensembldb.ensembl.org:欧洲服务器,只有该服务器可访问 GRCh37 数据集
useastdb.ensembl.org:美洲服务器
asiadb.ensembl.org:亚洲服务器
martdb.ensembl.org:提供对 BioMart 数据库的公共访问
这里提供了数据库匿名访问权限,不同于网页端僵硬的访问操作,数据库中我们可以更灵活地获取更细粒度的信息。
Ensemble 用到的数据库管理系统包括为人熟知的 MySQL 和 MariaDB。
这里说个小故事,便于理解 MySQL 和 MariaDB 的渊源:
MySQL 率先由麦克尔·维德纽斯主导开发,后来以 10 亿美元卖给了 SUN 公司。SUN公司就是那个拥有 java 语言版权的大佬,但经营不善由甲骨文公司收购。甲骨文公司的产品 Oracle 本身也是数据库管理软件,和 MySQL 有竞争。收购后,MySQL 开发社区马上意识到他们的软件 MySQL 有潜在闭源风险。于是,迈克尔率领社区成员构建新分支,并以自己女儿的名字玛利亚命名,即 MariaDB。
总的来看,二者师出同门,在使用方面方面并无本质区别。在存储引擎方面,MariaDB 10.0.9版起使用XtraDB 代替了 MySQL 的 InnoDB。
依据下面的请求信息,我们就可以登录 Ensemble 的数据库服务器。
| Server | User | Password | Port(s) | Version | Notes |
|---|---|---|---|---|---|
| ensembldb.ensembl.org | anonymous | - | 3306 & 5306 | MySQL 5.6.33 | From Ensembl 48 onwards only |
| useastdb.ensembl.org | anonymous | - | 3306 & 5306 | MariaDB 10.0.30 | Current and previous Ensembl version only |
| asiadb.ensembl.org | anonymous | - | 3306 & 5306 | MariaDB 10.0.30 | Current and previous Ensembl version only |
| martdb.ensembl.org | anonymous | - | 5316 | MariaDB 10.0.30 | From Ensembl 48 onwards only |
| ensembldb.ensembl.org | anonymous | - | 3337 | MySQl 5.6.33 | Databases for archive GRCh37 - release 79 onwards |
| ensembldb.ensembl.org | anonymous | - | 4306 | MySQL 4.1.20 | Up to Ensembl 47 only |
| martdb.ensembl.org | anonymous | - | 3316 | MySQL 4.1.20 | Up to Ensembl 47 only |
二、连接数据库服务器
1、这里使用 Navicat 作为连接工具,下载地址:
https://www.navicat.com/en/download/navicat-premium
2、左上角使用 MySQL 连接
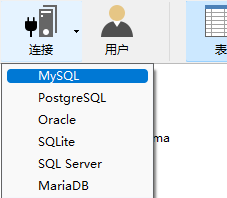
3、填写连接信息
在国内推荐填写亚洲服务器地址,提高访问速度
4、打开连接,即可看到其中的数据库
5、打开人类最新数据库
数据库命名规则:物种拉丁名_功能库名_架构版本_基因组版本
比如:homo_sapiens_core_104_38
代表:人类 hg38基因组的 Ensembl 104 版本架构数据库
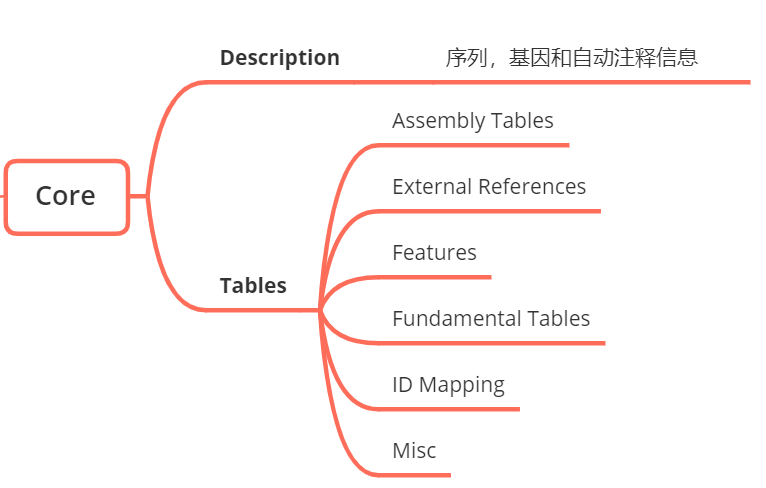
三、Ensemble Core 数据库维度模型
这里我们以 Ensemble 的核心数据库为例,来简单看下它的架构,作为 Ensemble 最基本的库,它承担着序列,基因,与注释信息的记录任务。
为了方便理解,从逻辑上分为六种数据表:
Assembly Tables
External References
Features
Fundamental Tables
ID Mapping
Misc
如下图:
我们以其中最重要的 Fundamental Tables 为例,从数据维度模型的角度,由易到难来看看 Ensemble 是怎样组织数据的,便于我们今后利用其进行研究工作。
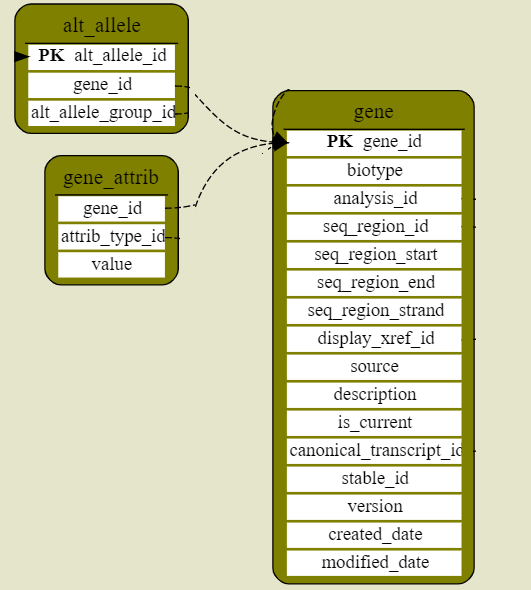
1、星型
这应该是最基本的维度模型,由简单事实和维度表构成。
以事实表为核心,所有维度度表关联到事实表,呈现星型分布。一般是构建数据库的基础模型。下图中展示了简化后的 Fundamental Tables 的 ER 图。
gene
该表记录有关基因名,序列类型,序列起止等信息,以 gene_id 为主键
alt_allele
可能是直系同源的等位基因的信息存储在这里,与 gene 表以 alt_allele_id 连接
gene_attrib
用于启用基因属性表
可以看到所有基因信息并不是用一张表存储,而是用主键,外键链接,用于降低冗余,符合范式要求
2、雪花型
在星型基础上,进一步分解维度表,有点“套娃维度表”那味儿。
但是,这种模型会增加代码量,维护成本高,性能也差,尤其使用 Hadoop 时,性能差距会加大,因此如果做库时一般不推荐单独使用。
这里的表同样与上述结构类似,由于等位基因还需要更多维度的信息,因此加入一个新表用于解释 alt_allele 表。
alt_allele_attrib
这里会标注了等位基因的所有不同属性,增加了 alt_allele 信息维度,使信息更清晰全面。

3、星系型
多个事实表的雪花型的组合。
特征就是多个事实表会共享维度表,也是构建数据库的常用模型。
在这里利用上面的逻辑理一理,大概就能看懂下图:
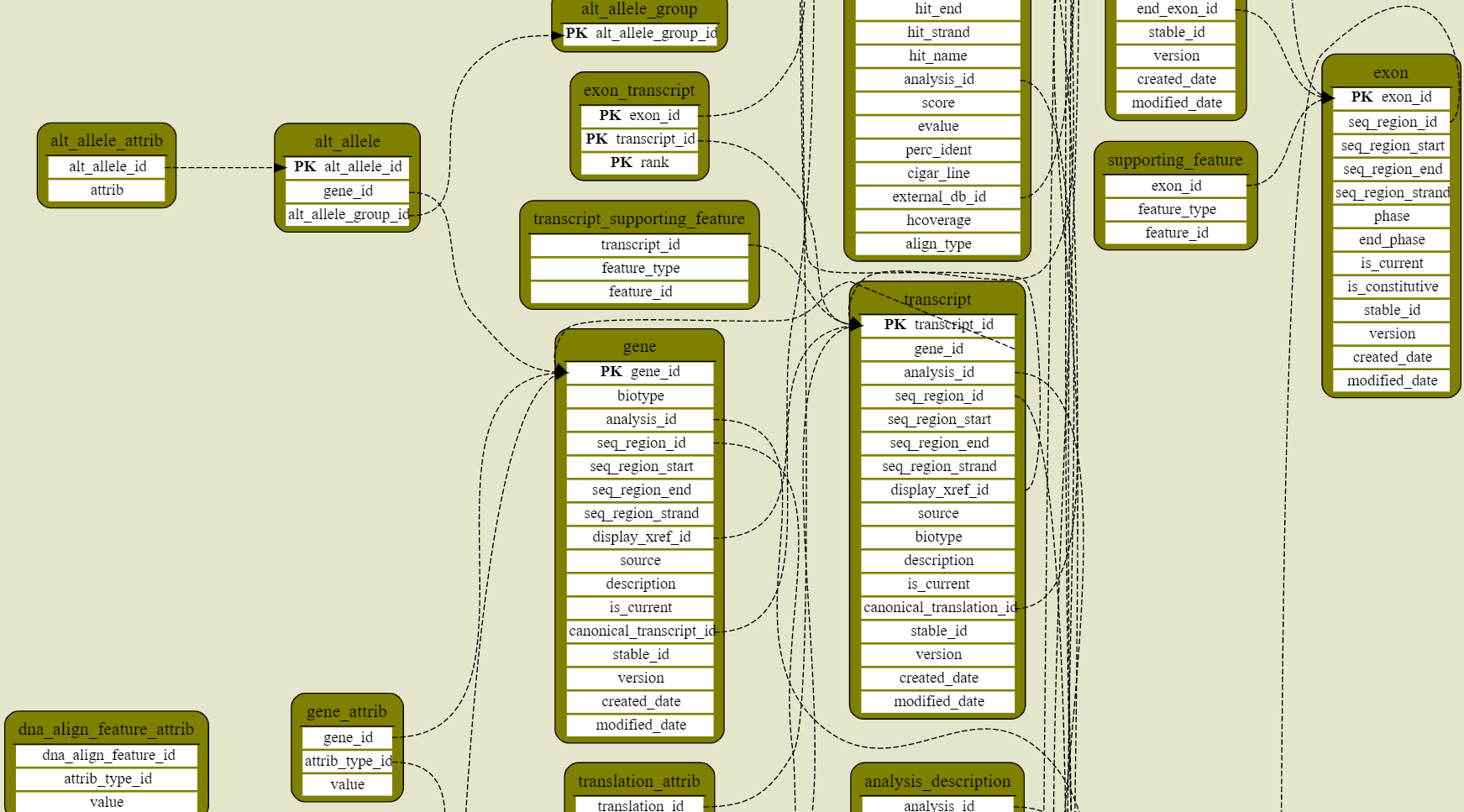
而实际上,Ensemble 也就是使用这样的星系型维度模型构建。
对于生信数据分析人员来说,使用时只要找到我们关心的事实表,依次找到维度表,从而快速拿到全面,清晰的即时数据,便于我们用到今后的生信数据分析工作中。
更多生信数据库知识会更新于:https://zhenglei.blog.csdn.net/










