最新 NCBI 上传测序数据教程 (图文详解)
在做完测序后,一般期刊会要求提供公共数据ID,可以上传的数据库可以是 NCBI,EBI,DDBJ,以及国内的 NGDC。今天我们以 NCBI 上传数据为例。
0、基础知识
NCBI 的其中一个数据库 GEO 用于管理和存储我们的测序数据。GEO 构建之初用于存放芯片数据,后来成为随着测序技术的快速发展,逐渐成为高通量测序数据的主要存储库之一。
其中每个项目都可以包含有测序后原始数据信息,如 sra,fastq 等文件信息等,以及上游分析后的数据,如count,bam,bw 等文件。
需要注意的是上面提到的原始数据是存储的文件信息,真正的 raw data 文件是由 Sequence Read Archive (SRA) 库来管理。接下来的工作就是去 SRA 上传我们的数据。
1、登录或注册用户
网址:https://www.ncbi.nlm.nih.gov/account/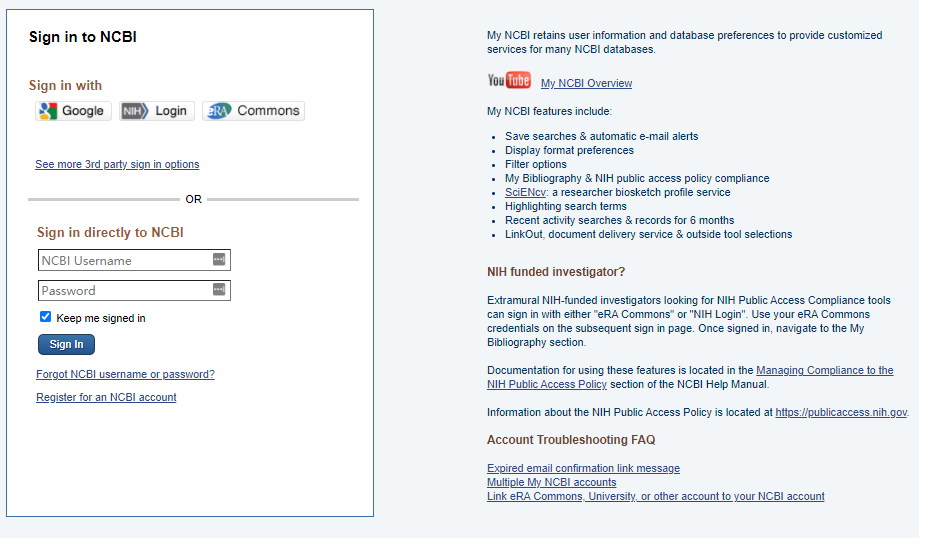
2、进入SRA
网址:https://submit.ncbi.nlm.nih.gov/
向下滚动,找到Sequence Read Archive (SRA)工具,点击Submit
2、新建提交
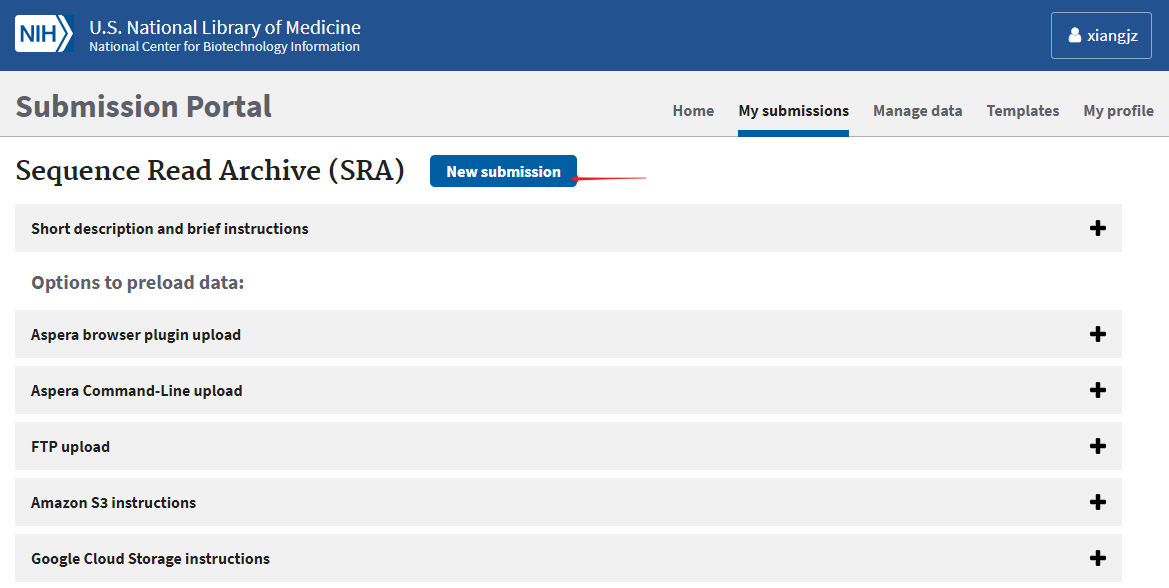
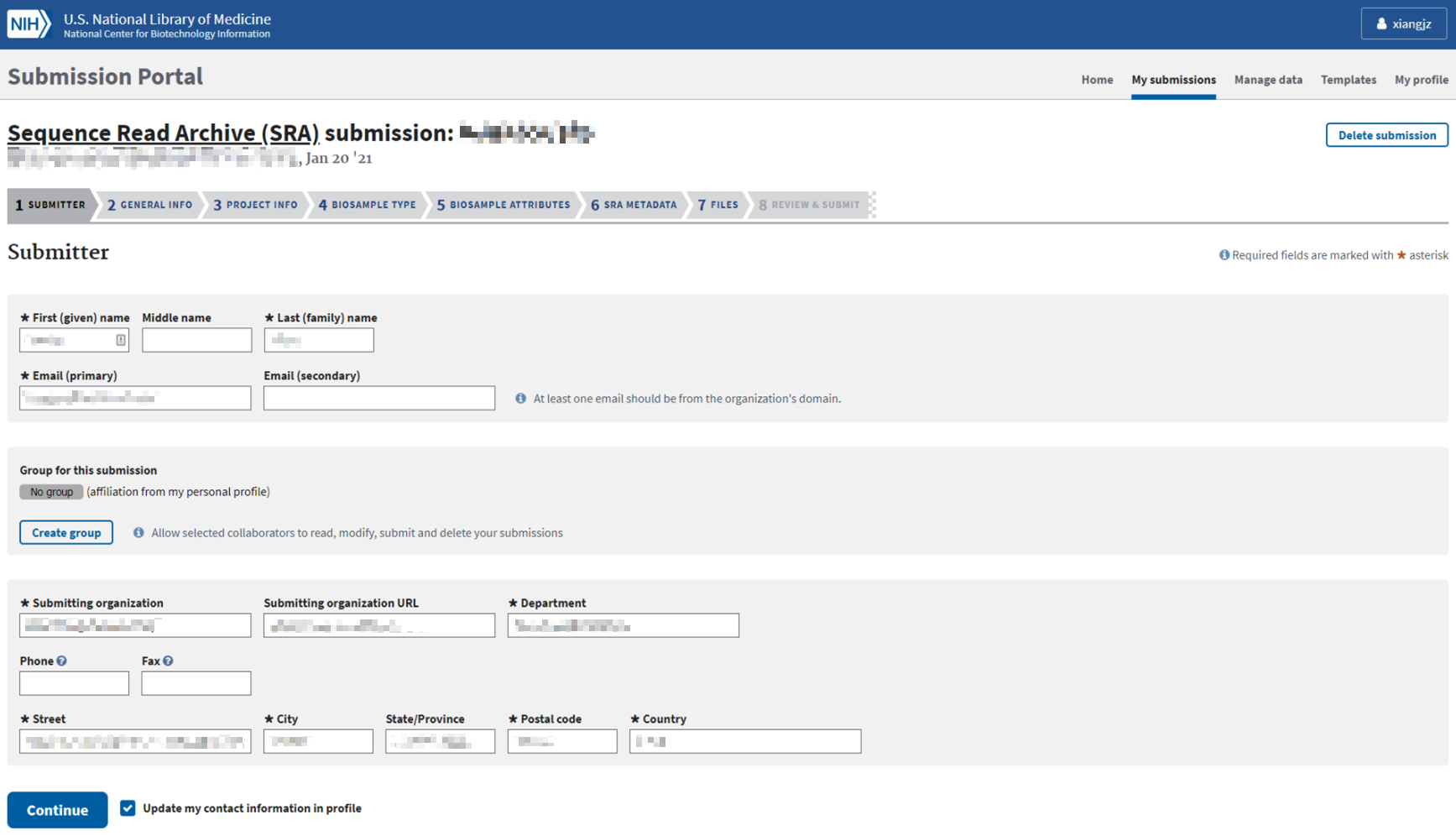
3、按要求填写信息
这一步需要的信息比较多,建议下载表格后,直接上传即可。
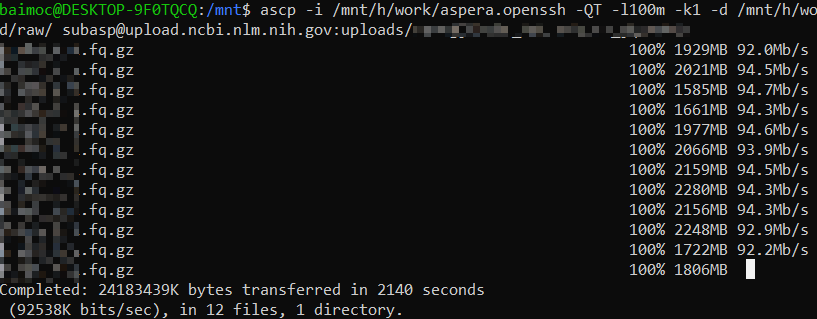
4、使用ascp
可以使用 ftp 上传,但 NCBI 提供了更为快速的上传工具 aspera ,这里实测是在 100 Mb/s 左右浮动,安装参考:https://blog.csdn.net/u011262253/article/details/107190684
希望在不久的将来,我们国家 NGDC 的 GSA 数据库也能有类似的工具,而不是只提供 ftp 上传。
|

5、等待上传完成
6、确认是否上传完成
重复以上命令,ascp 会检测文件上传是否完整,如果上传完成,会直接 skipped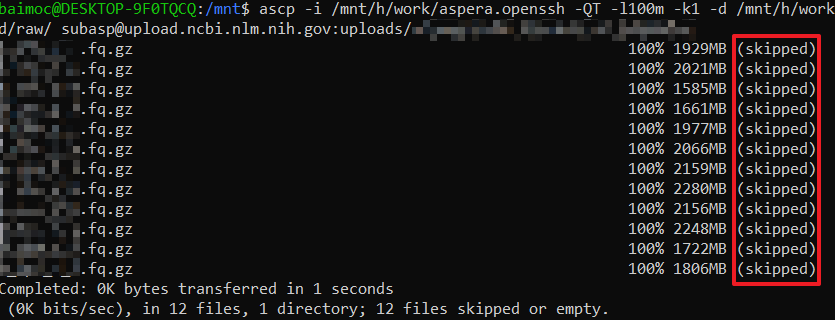
7、检测上传数据是否正确
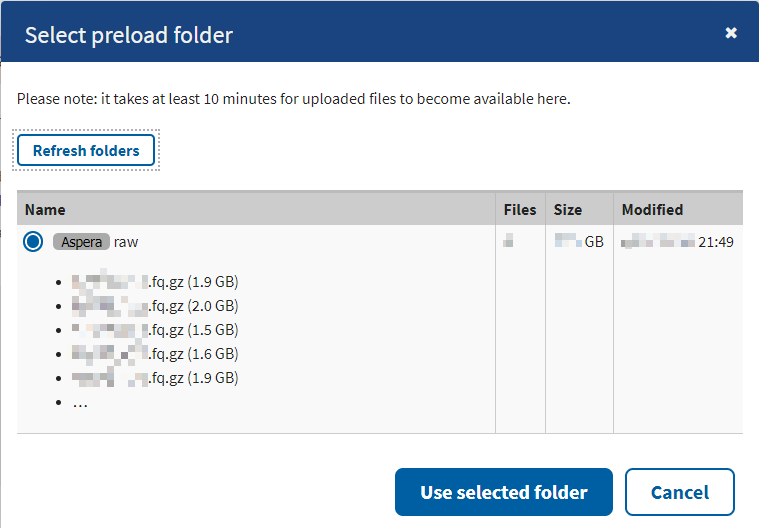
确认无误后,确定公开时间等信息,等待审核通过后,会获得官方提供的唯一 GSE 标识符。
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来自 白墨!
评论








